Metagenoma shotgun
La secuenciación completa de genomas microbianos desde matrices complejas (shotgun metagenome), tales como virus, bacterias o plásmidos, puede ser útil para ensayos de calidad de alimentos en salud pública, la vigilancia de enfermedades infecciosas y estudios de epidemiología molecular, como también para estudios de metagenómicas ambientales.
Especificaciones del Servicio
Requerimientos de las muestras:
- Concentración mínima: 8 ng/uL de DNA
- Volumen mínimo: 20 uL
Secuenciación:
- Plataforma Illumina HiSeq, lecturas 100 o 150 Pair End, ~ 5Gb por muestra
Análisis Bioinformático
- Control de calidad de las lecturas y limpieza de adaptadores
- Prueba de ensambladores
- Enmascaramiento de zonas repetitivas
- Refinamiento de los ensambles
- Estadística del ensamble
- Predicción de genes
- Anotación de genes codificantes
- Anotación de genes no codificantes
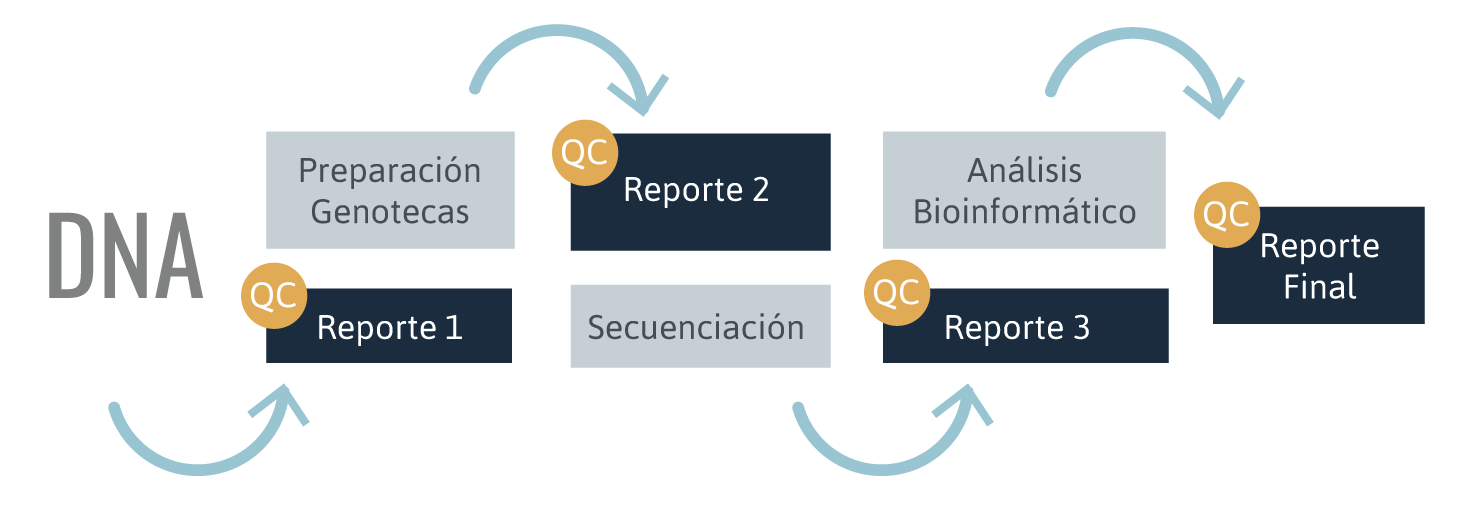
Durante las distintas etapas del servicio, Ud. recibirá:
- Reporte QC muestras
- Reporte genotecas
- Reporte secuenciación
- Raw data
- Data procesada y anexos
- Reporte Final
¿Tiene usted alguna otra necesidad que no hayamos cubierto?
Servicios
- Secuenciación NGS
- Bioinformática
- Ensamble y Anotación de Genomas
- ddRAD
- Análisis de datos de Genotyping by Sequencing (GBS)
- Análisis de datos de Exomas
- Identificación de variantes de amplicones
- Análisis de datos de RNA-Seq
- Análisis de datos de smRNA
- Análisis de datos de dual RNA-Seq
- Análisis taxonómico de 16S
- Análisis taxonómico de 18S/ITS
- Análisis de datos metagenómicos
- Análisis de datos metatranscriptómicos
- Análisis de datos de Deep-sequencing viral