Análisis 16S rRNA
El RNA ribosomal 16S (o 16S rRNA) es un componente de la subunidad pequeña de los ribosomas de bacterias y arqueas. El gen que codifica para el 16S rRNA permite la exploración de la diversidad microbiana de una comunidad sin la necesidad de cultivar las bacterias en el laboratorio. El análisis de comunidades microbianas mediante la secuenciación del 16S, permite la identificación de forma eficiente de la diversidad microbiana, sin importar el origen de la muestra, la que puede ser ambiental (suelos, agua, superficies, etc) o provenir de otro organismo (microbioma).
Especificaciones del Servicio
Requerimientos de las muestras:
- Concentración mínima: ≥ 6 ng/uL de DNA
- Volumen mínimo: 20 uL
Secuenciación:
- Plataforma Illumina MiSeq 150PE e Illumina HiSeq 250PE. Entre 10.000 – 100.000 reads/muestra dependiendo de condiciones de secuenciación.
Análisis Bioinformático
- Control de calidad de las lecturas y limpieza de adaptadores
- Desmultiplexar muestras
- Filtro de secuencias quimeras
- Control del número mínimo de reads por muestra
- Análisis de diversidad
- Generación de gráficos
- Análisis de taxas diferenciales (Solo en caso que las muestras se puedan tratar como replicas)
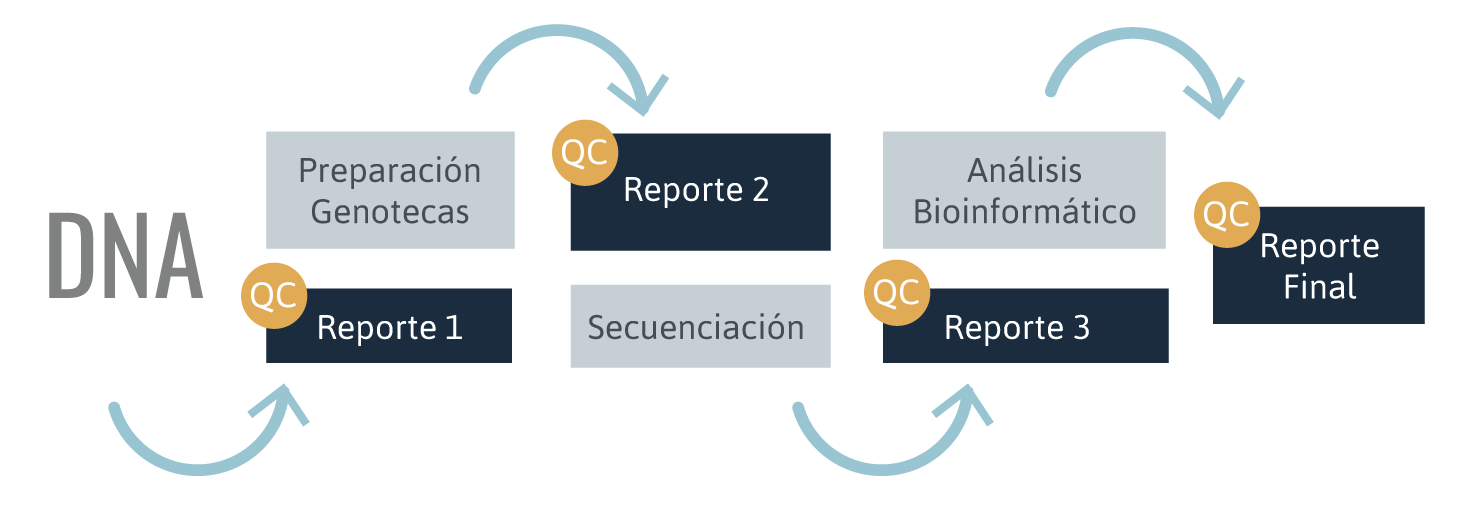
Durante las distintas etapas del servicio, Ud. recibirá:
- Reporte QC muestras
- Reporte genotecas
- Reporte secuenciación
- Raw data
- Data procesada y anexos
- Reporte Final
¿Tiene usted alguna otra necesidad que no hayamos cubierto?
Servicios
- Secuenciación NGS
- Bioinformática
- Ensamble y Anotación de Genomas
- ddRAD
- Análisis de datos de Genotyping by Sequencing (GBS)
- Análisis de datos de Exomas
- Identificación de variantes de amplicones
- Análisis de datos de RNA-Seq
- Análisis de datos de smRNA
- Análisis de datos de dual RNA-Seq
- Análisis taxonómico de 16S
- Análisis taxonómico de 18S/ITS
- Análisis de datos metagenómicos
- Análisis de datos metatranscriptómicos
- Análisis de datos de Deep-sequencing viral